![]() Index aliphatique
Index aliphatique
L'index aliphatique (Ikai A.) est défini comme le volume relatif des chaînes latérales des aminoacides aliphatiques (alanine, valine, isoleucine et leucine) d'une protéine., calculé de la manière suivante :
Index aliphatique= P(Ala) + a * P(Val) + b * ( P(Ile) + P(Leu) )
où P(aa) est la fraction molaire en pourcentage de l'aminoacide considéré et où a et b sont des coefficients de poids (a=2,9 et b= 3,9).
On considère que la valeur de cet index aliphatique rend compte de la stabilité de la protéine vis à vis de la température.
![]() Coefficient d'extinction à 280nm
Coefficient d'extinction à 280nm
Les coefficients d'extinction molaire (CEM) de la protéine sont calculés d'après Gill and Von Hippel (en présence de chlorure de guanidine 6M à pH 6,5). La relation est alors la suivante :
CEM = (a * ETyr) + (b * Etrp) + (c * ECys) , où a, b et c sont respectivement le nombre de résidus, et Eaa leurs coefficients d'extinction molaire.
A 280nm, ils sont respectivement égaux à : ETyr=1280 ETrp=5690 ECys=120
A 280nm le coefficient d'absorption d'une solution à 0,1% est :
Abs 1% = CEM / MM MM : masse moléculaire
![]() Remarques :
Remarques :
Le programme calcule deux valeurs de CEM et Abs 0,1% :
- l'une en ne prenant pas en compte les cystéines
- l'autre en prenant en compte toutes les cystéines
![]() Point isoélectrique
Point isoélectrique
La valeur du pH correspondant au point isoélectrique (charge nette moyenne nulle) est calculée à l'aide de la théorie des équilibres multiples (Wyman ou encore Bellon).
Nous considérons que les différents groupements ionisables sont tous indépendants l'un vis à vis de l'autre (expérimentalement, nous pouvons nous en approcher en présence d'urée 6M) et nous cherchons les équilibres partiels qui encadrent la forme de la protéine dont la charge nette moyenne est nulle. A l'aide des constantes successives de dissociation cela peut se représenter sous la forme (en partant de la forme la plus acide) :
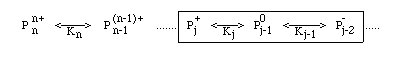
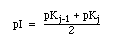
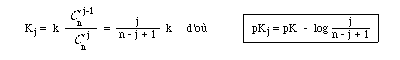
![]() Remarques :
Remarques :
Le programme calcule deux valeurs de pI,
- l'une (classique) sans tenir compte des cystéines
- l'autre, en prenant en compte les cystéines (groupements sulfhydryles libres)
![]() Courbe de titrage
Courbe de titrage
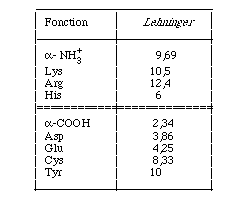
Les résidus portant une fonction dissociable, ionisable sont les suivants (les pK sont tirés du "Lehninger") :

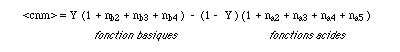
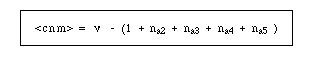
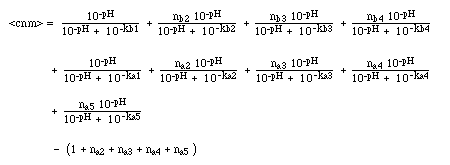
![]() Comment récupérer le graphe
Comment récupérer le graphe
Si vous n'y arrivez pas à l'aide du menu contextuel : lorsque le pointeur de la souris est dans le graphe, il change de forme et vous indique un lien vers le fichier de type "JPEG". Cliquez et cela entrainera l'ouverture d'une nouvelle page avec uniquement l'image que vous pouvez sauvegarder à l'aide de l'item "Save as" du menu "File". Le fichier est de type "jpg" et vous avez sûrement un logiciel pour le traiter ou le transformer dans un autre type.
- Ikai A. J. Biochem. 88:1895-1898(1980) - Gill S. and Von Hippel P. Anal. Biochem. 182, 319-326 (1989) - Wyman J. and Stanley J. Binding and linkage Ed : University Science Books (1990) - Bellon B. Equilibres Multiples (Cours : p 34 et 35) (1994) * - Lehninger A. Biochimie Ed : Flammarion (1972) - Bellon B. Equilibres Multiples (Corrigés : n° 15) (1994) * - * Un clin d'oeil à Michel Delaage
![]()
ABIM : © Ixe et Ygrec : ce programme utilise le programme "fly" qui crée des images de type GIF à la volée
Thanks to Martin Gleeson and credit to the University of Melbourne and the Quest Protein Database Center (Cold Spring Harbor Labs)